Các phản ứng liên quan đến việc vật chủ sản xuất các đoạn DNA sao chép virus (vcDNA) từ RNA virus ở cả dạng tuyến tính (lvcDNA) và dạng vòng (cvcDNA) thông qua enzyme sao chép ngược của vật chủ nội sinh (RT). Tiếp theo là sản xuất RNA can thiệp nhỏ (siRNA) và một Các phản ứng can thiệp RNA (RNAi) của tế bào và hệ thống, cùng với việc tự động chèn một số đoạn vcDNA vào DNA bộ gen của vật chủ dưới dạng các yếu tố virus nội sinh (EVE). Nếu EVE tạo ra RNA antisense, chúng cũng có thể tạo ra phản ứng RNAi bảo vệ tế bào có thể di truyền.
Gần đây người ta đã tiết lộ rằng EVE của tôm cũng có thể tạo ra cvcDNA có thể được phân lập và giải trình tự có chọn lọc như một phương tiện sàng lọc EVE trong đàn tôm giống. Dựa trên dữ liệu từ toàn bộ trình tự bộ gen của tôm sú Penaeus monodon, người ta đã chứng minh rằng EVE xuất hiện dưới dạng các mảnh cùng với các chuỗi yếu tố có thể thay thế của vật chủ trong các cụm giống gen piRNA của tôm chủ được bao quanh bởi các đoạn lặp tuyến tính.
Các đoạn bộ gen virus trong các cụm này bị xáo trộn về hướng đọc và vị trí bộ gen ban đầu và cũng có thể trùng lặp quá mức khi ánh xạ tới bộ gen virus mục tiêu. Do đó, các trình tự đọc ngắn từ các quy trình lắp ráp và giải trình tự thông lượng cao thông thường có thể dẫn đến trình tự toàn bộ bộ gen virus có vẻ liên tục, hoàn chỉnh hoặc gần như hoàn chỉnh khi được ánh xạ dựa trên cơ sở dữ liệu về các trình tự virus đã biết.
Nghiên cứu mới đây của Cơ quan Phát triển Khoa học và Công nghệ Quốc gia Thái Lan (2024) được thực hiện nhằm đánh giá cvcDNA của WSSV có thể được chiết xuất từ một giống tôm thuần hóa được chọn lọc để có khả năng kháng WSSV và nó có thể được giải trình tự để sử dụng trong việc thiết kế các mồi để phát hiện PCR WSSV-EVE ở từng cá thể tôm.
Các mẫu DNA (n=50) trong ethanol 95% từ đàn tôm thẻ chân trắng được lấy từ Tập đoàn Nuôi trồng Thủy sản Quốc gia (NAQUA), Vương quốc Ả Rập Saudi. Đàn tôm giống này nằm trong một trung tâm NTTS an toàn sinh học không có WSSV và vẫn không có WSSV cho đến nay (thời gian 10 năm). Đàn có nguồn gốc từ những cá thể sống sót sau đợt bùng phát bệnh WSD đã được sàng lọc để tránh WSSV lây nhiễm và tạo thành một đàn không chứa mầm bệnh.
Quá trình trích xuất DNA được thực hiện bằng cách sử dụng bộ chiết DNA mini tế bào Exgene™ SV (GeneAll, Hàn Quốc) và nồng độ DNA được xác định bằng máy quang phổ Nanodrop (ThermoFisher Scientific, Hoa Kỳ). Lượng DNA gốc riêng lẻ được pha loãng đến 50ng/µl để phân tích PCR và 2µg từ mỗi mẫu được sử dụng cho bước tách DNA vòng (cirDNA).
Sàng lọc PCR sơ bộ được thực hiện riêng lẻ với một phần của tất cả 50 chiết xuất DNA bằng cách sử dụng các đoạn mồi được thiết kế để phát hiện sự hiện diện của WSSV-EVE366 như đã mô tả trước đây. EVE366 trước đây được báo cáo là EVE chiếm ưu thế (84,8%) ở tôm sú, đàn giống của Trung tâm cải tiến di truyền tôm ở Thái Lan. Các tác giả đã đưa ra giả thuyết rằng EVE366 có thể mang lại một số lợi thế có chọn lọc. Các chiết xuất DNA từ 50 con tôm trong nghiên cứu này đã được sàng lọc để phát hiện sự hiện diện của EVE366 trước khi DNA được gộp lại và được phân lập và giải trình tự cirDNA.
Tổng cộng 10 trong số 28 mẫu DNA có kết quả xét nghiệm dương tính với EVE366 đã được chọn và gộp lại để chiết xuất cvcDNA cũng như chuẩn bị cho việc giải trình tự DNA như mô tả trước đây (Hình 1). Tóm lại, hai microgam DNA bộ gen gộp của tôm đã được chia mẫu và được tiêu hóa DNA tuyến tính bằng cách sử dụng DNase an toàn với plasmid trong 4 ngày. CirDNA còn lại được kết tủa bằng ethanol-acetate và sau đó nồng độ được xác định bằng Máy đo huỳnh quang Qubit.
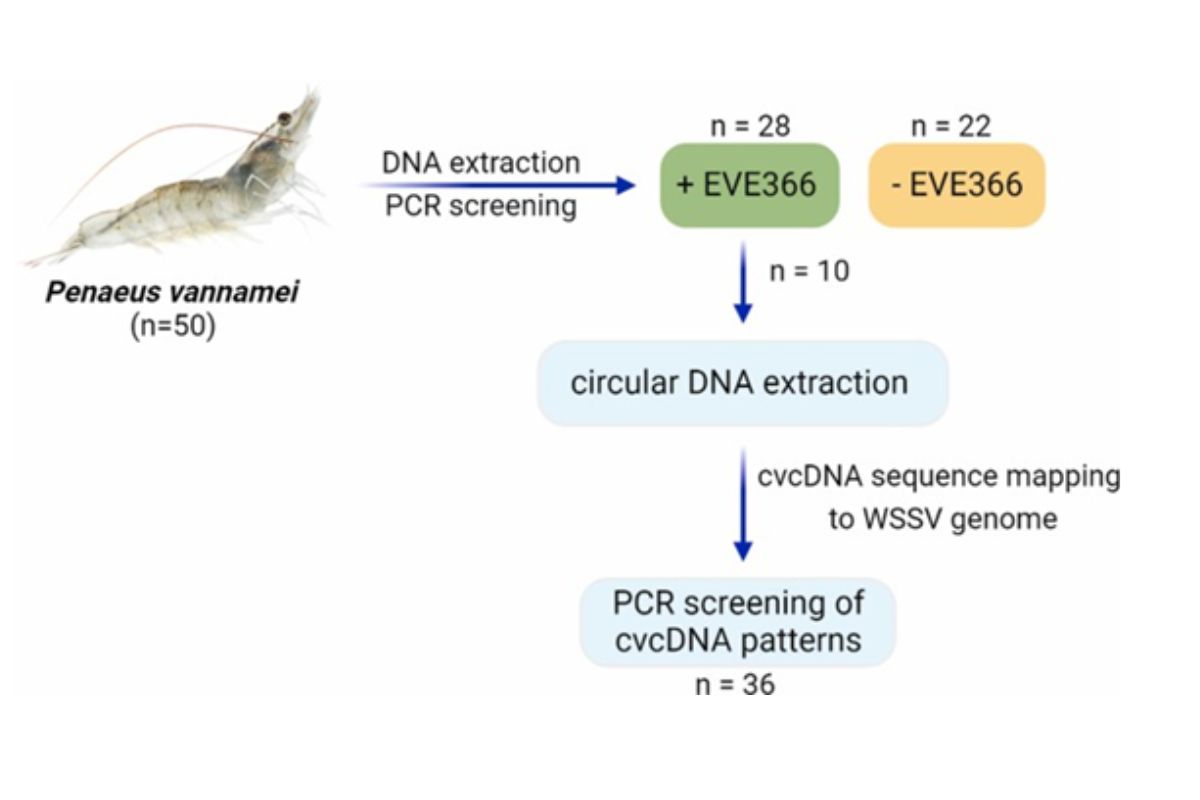 Hình 1. Sơ đồ thể hiện kế hoạch phân lập, giải trình tự và phân tích các mẫu cDNA giả định có nguồn gốc từ 50 mẫu tôm bố mẹ
Hình 1. Sơ đồ thể hiện kế hoạch phân lập, giải trình tự và phân tích các mẫu cDNA giả định có nguồn gốc từ 50 mẫu tôm bố mẹ
Sàng lọc sơ bộ EVE366 trong các mẫu tôm bố mẹ NAQUA
Một bộ gồm 50 mẫu DNA từ đàn giống tôm thẻ chân trắng NAQUA được chứng minh là không có WSSV đã được xét nghiệm WSSV-EVE366 trước đây được phát hiện là loại phổ biến nhất trong số 4 EVE được biết là xuất hiện ở tôm sú từ Thái Lan. Mồi chuyển tiếp WSSV-EVE366 nằm ở vị trí 251.608 đến 251.635 trong bản ghi GenBank AF440570.
Đây là thử nghiệm sơ bộ dựa trên giả định rằng các mẫu DNA dương tính với PCR có thể chứa WSSV-cvcDNA làm chỉ báo cho sự hiện diện của WSSV-EVE366. Bước đầu tiên, tất cả 50 mẫu chiết xuất DNA đều được tiến hành xét nghiệm PCR để tìm sự hiện diện của DNA ty thể như một biện pháp kiểm soát tích cực đối với sự hiện diện của DNA vòng nguyên vẹn trong dịch chiết (Hình 2A).
Khi các mẫu tương tự cũng được kiểm tra PCR để tìm sự hiện diện của WSSV-EVE366, 28 trong số 50 mẫu DNA đã cho kết quả dương tính (Hình 2B). Những kết quả này đủ đáng khích lệ để tiếp tục trích xuất cvcDNA, sau đó là giải trình tự tất cả các mẫu trong lô, mỗi mẫu có 10 mẫu. Kết quả xét nghiệm PCR WSSV-EVE366 cho tất cả các mẫu được thể hiện dưới dạng bảng trong Bảng 2.
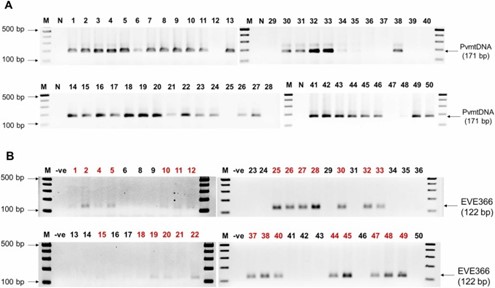
Hình 2. Xét nghiệm PCR sơ bộ với 50 mẫu DNA được chiết xuất
A. Ảnh hiển vi đảo ngược trên gel agarose cho thấy kết quả xét nghiệm PCR dương tính với sự hiện diện của DNA ty thể hình tròn của tôm trong tất cả 50 mẫu.
B. Ảnh vi mô đảo ngược của các dải khuếch đại PCR cho thấy sự hiện diện của trình tự mục tiêu WSSV-EVE366 (số màu đỏ) đối với 28 trong số 50 mẫu DNA.
Các kết quả thu được về sự hiện diện của DNA ty thể (đại diện DNA vòng tròn của vật chủ) và sự hiện diện của EVE366 trong tất cả 28 mẫu đã khuyến khích chúng tôi tiếp tục trích xuất cvcDNA để gộp lại và gửi đi phân tích NGS. Đối với nhóm chiết xuất DNA đầu tiên, chúng tôi đã chọn 10 chiết xuất đầu tiên (đường viền màu xám trong Bảng 2) cho kết quả xét nghiệm PCR dương tính với WSSV-EVE366.
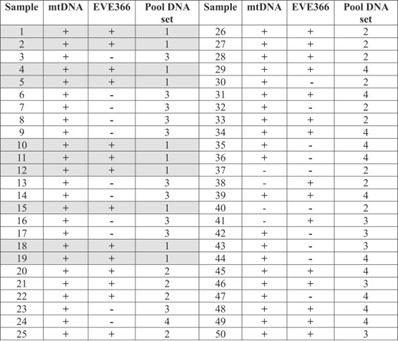
Bảng 2. Bảng tóm tắt thể hiện đặc điểm nhận dạng và đặc điểm của 50 mẫu được xét nghiệm mtDNA và EVE366
Khi các trình tự đọc ngắn từ các mẫu được căn chỉnh với toàn bộ trình tự bộ gen WSSV của một loại WSSV gây chết người còn tồn tại (bản ghi GenBank AF440570), những trình tự có trình tự tương đồng cao với WSSV (99–100%) được ánh xạ tới các vị trí nằm trên toàn bộ WSSV bộ gen gồm 307, 287 cặp bazơ (bp) (Bảng 3 & Hình 3). Bây giờ chúng ta biết rằng các trình tự của các nhánh WSSV rất có thể là kết quả nhân tạo của quá trình tập hợp trình tự sử dụng các trình tự đọc nhỏ chồng chéo được tập hợp theo trình tự tham chiếu, chứ không phải là trình tự liền kề thực tế trong bộ gen mục tiêu.
Điều này đã được bộc lộ rõ ràng đối với EVE của virus tôm IHHNV trong trình tự bộ gen tôm đầy đủ được phát triển bằng công nghệ đọc dài. Các đoạn EVE của bộ gen IHHNV xuất hiện thành các cụm được xáo trộn (về vị trí và hướng đọc), một số có trình tự chồng chéo dẫn đến tổng độ dài trình tự IHHNV-EVE tích lũy vượt quá toàn bộ chiều dài bộ gen IHHNV.
Khi nhìn vào Hình 4, điều quan trọng cần lưu ý là các lần truy cập tần số thấp được tìm thấy phân bố trên toàn bộ bộ gen của WSSV (Hình 3). Ngoài ra, Hình 4 không đưa ra dấu hiệu nào về cách phân bổ các mảnh được sử dụng để chuẩn bị bản đồ này trong bộ gen của 10 cá thể tôm riêng lẻ được sử dụng để chuẩn bị nhóm DNA.
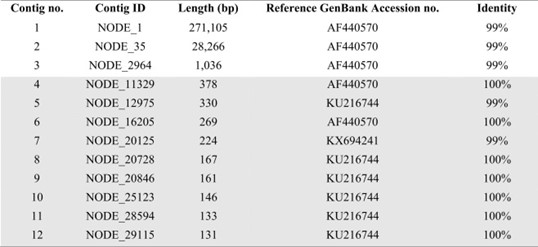
Bảng 3. Danh sách các trình tự được tập hợp từ bộ mẫu cvcDNA gộp 1
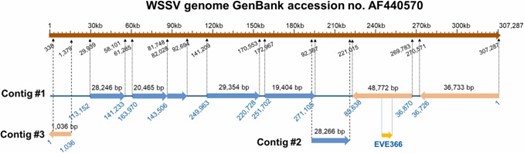
Hình 3. Bản đồ vị trí của các nhánh khác nhau được xây dựng từ dữ liệu NGS và hiển thị sự phân bố trên toàn bộ bộ gen của WSSV đối với các trình tự axit nucleic có tính tương đồng cao
Kết quả về các đoạn đọc tần số cao được ánh xạ tới một phần nhỏ của bộ gen WSSV
Hầu hết các đoạn cvcDNA có tính tương đồng cao với WSSV xảy ra ở mức phổ biến thấp, trong khi các đoạn đọc tần số cao (HFRF) chỉ ánh xạ tới một phần nhỏ của bộ gen bao phủ khoảng 1400 bp (Hình 4A và B). Do đàn giống nguồn được phát triển từ những cá thể sống sót sau đợt bùng phát dịch bệnh WSSV chưa bị nhiễm bệnh nên dữ liệu cho thấy các chuỗi EVE tần số cao từ vùng đó của bộ gen WSSV có thể đã được chọn lọc tự nhiên ở những cá thể sống sót sau đợt bùng phát dịch ban đầu và sau đó tiếp tục trong các thử nghiệm thử thách được thiết kế để chọn lọc tôm có khả năng chống chịu tốt nhất với thách thức WSSV.
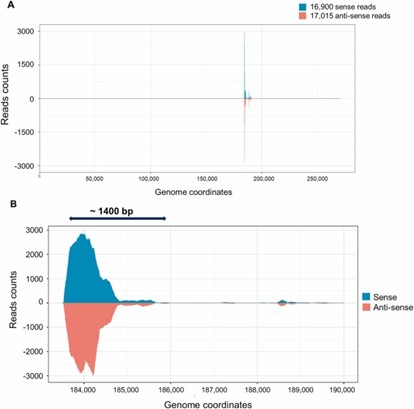
Hình 4. Các đường viền khác nhau được xây dựng từ dữ liệu NGS
Kết quả về biến thể EVE cá thể tôm cao được tiết lộ qua phân tích HFRF-PCR
Dựa trên giả thuyết rằng các đoạn đọc tần số cao (HFRF) cho thấy sự lựa chọn tích cực do khả năng chịu WSSV gây ra ở các chất mang, các bộ mồi PCR được thiết kế (Bảng 1) để bao phủ vùng 1400 bp như trong Hình 4. Chúng được thể hiện dưới dạng sơ đồ được so sánh với trình tự tham chiếu GenBank AF440570 trong Hình 5.

Hình 5. Sơ đồ các bộ khuếch đại dự kiến sử dụng bộ mồi được trình bày trong Bảng 1.
Trong Hình 4, bộ gen của bản ghi GenBank AF440570 đã được sử dụng làm khuôn mẫu để thiết kế các đoạn mồi được minh họa trong Hình 5. Trên thực tế, các bộ khuếch đại thu được bằng cách sử dụng DNA bộ gen từ các mẫu tôm NAQUA có thể khác nhau. Vì vậy, tất cả các bộ khuếch đại như vậy sẽ phải được sắp xếp theo trình tự riêng lẻ. Cũng lưu ý rằng các bộ Primer 2–4 đều nằm trong phạm vi bản đồ được bao phủ bởi bộ Primer 1.
Do đó, nếu một bộ khuếch đại thu được bằng cách sử dụng bộ Primer 1 với mẫu tôm phát sinh từ một EVE liên tục, duy nhất khớp chính xác với trình tự của AF440570, nó sẽ bao gồm trình tự của tất cả các bộ mồi còn lại và do đó sẽ cho kết quả xét nghiệm PCR dương tính đối với cả 4 bộ mồi. Mặt khác, chẳng hạn, kết quả xét nghiệm PCR dương tính chỉ riêng với bộ mồi 2 sẽ không cho thấy sự hiện diện của các trình tự liền kề liên quan đến các trình tự được nhắm mục tiêu bởi các bộ mồi khác.
Trong số 50 mẫu tôm ở Bảng 2 trên, chỉ còn lại 36 mẫu có đủ DNA để thực hiện phản ứng PCR với cả 4 bộ mồi. Khi thực hiện điều này với tất cả 36 mẫu (144 phản ứng), kết quả nêu trong Bảng 4 thu được và được tóm tắt bằng hình ảnh trong Hình 6. Từ Bảng 4, có thể thấy rằng chỉ 4 trong số 36 mẫu cho kết quả xét nghiệm âm tính khi sử dụng 4 phương pháp PCR và 32 trong số 36 con tôm (89%) cho kết quả xét nghiệm PCR dương tính với ít nhất một trong các đoạn trình tự WSSV được nhắm mục tiêu bởi 4 bộ mồi.
Mẫu này tương tự với mẫu thu được khi sử dụng các lớp mồi chồng lên nhau để sàng lọc 40 con tôm đã được chọn bằng phương pháp phát hiện virus IHHNV do WOAH khuyến nghị. Chỉ có 3 trong số 40 mẫu tôm (7%) dương tính với IHHNV khi sử dụng tất cả các mồi chồng lên nhau và xác nhận nhiễm IHHNV. 37 trường hợp còn lại (93%) chỉ cho thấy các đoạn gen của IHHNV và do đó đã cho kết quả xét nghiệm dương tính giả đối với IHHNV bằng phương pháp duy nhất được khuyến nghị.
Chỉ một trong số 36 mẫu tôm (#42) mang EVE có trình tự tương ứng với toàn bộ vùng tần số cao 1365 bp được ánh xạ tới bộ gen WSSV trong Hình 4. Ngoài ra, ngoại trừ mẫu số 42 dương tính với cả 4 mẫu này. bộ mồi, việc 35 mẫu còn lại không cho kết quả dương tính với 1–3 trong số 4 bộ mồi đã cung cấp thêm sự xác nhận rằng những mẫu đó không bị nhiễm WSSV. Việc giải trình tự bộ khuếch đại từ mẫu số 42 và giải trình tự lựa chọn ngẫu nhiên các bộ khuếch đại từ các bộ mồi 2, 3 và 4 khác cho thấy rằng tất cả đều có mức độ nhận dạng trình tự rất cao (99–100%) đối với loại WSSV còn tồn tại.
Đặc biệt quan tâm là các bộ mồi HFRF cho kết quả xét nghiệm PCR dương tính với tỷ lệ cao trong số 36 con tôm được thử nghiệm (kết quả cuối cùng trong Bảng 4). Đó là bộ mồi 2 (25/36 = 69,4%) và bộ 3 (16/36 = 44%). Đây sẽ là mồi thử nghiệm sơ bộ được sử dụng để sàng lọc đàn giống nhằm thu được những cá thể phù hợp để sắp xếp các con lai nhằm kiểm tra khả năng bảo vệ của 2 EVE mục tiêu này.

Bảng 4. Kết quả xét nghiệm PCR sử dụng 4 bộ mồi với 36 mẫu từ đàn giống.
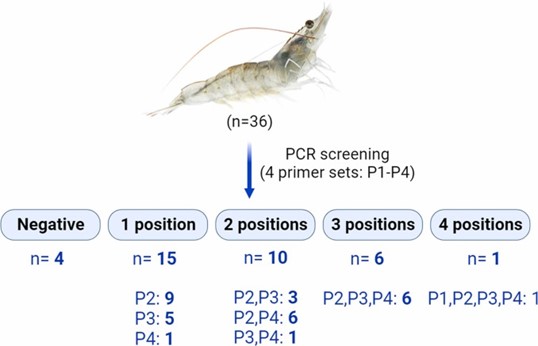
Hình 6. Hình ảnh kết quả xét nghiệm PCR sử dụng 4 bộ mồi với 36 mẫu từ đàn giống
Do đó, nghiên cứu này chỉ đơn giản mô tả bước đầu tiên trong quá trình phát hiện sự hiện diện của EVE đối với WSSV trong đàn giống SPF của tôm thẻ chân trắng nhằm chuẩn bị cho việc lựa chọn tôm bố mẹ mang nhiều tổ hợp HFRS EVE khác nhau để nhân giống. Sau khi nhân giống, con của các con lai khác nhau sẽ được phép phát triển đủ để cho phép gắn thẻ và thu thập các mẫu pleopod từ từng con tôm để chiết xuất và lưu trữ axit nucleic trước khi thử thách với WSSV. Sau thử thách WSSV, chúng tôi sẽ có thể đánh giá mối quan hệ giữa cấu hình biểu hiện EVE và khả năng sống sót hoặc tử vong. Hy vọng rằng quá trình này sẽ cho phép lựa chọn các đàn giống có khả năng chống chịu hoặc kháng bệnh WSSV được cải thiện.

_1772386127.png)







_1769663223.jpg)

_1769399561.jpg)
_1762138517.jpg)
_1772386127.png)





