Các MiRNA phát huy chức năng điều hòa của chúng bằng cách ghép cặp bazơ bổ sung với 3 vùng chưa được dịch mã (UTR) của bản phiên mã gen mục tiêu của chúng. Liên kết này hướng dẫn phức hợp im lặng do RNA tạo ra để phân tách hoặc kìm hãm RNA thông tin đích (mRNA). Do vai trò phổ biến của chúng trong việc điều hòa gen, miRNA tham gia vào nhiều quá trình sinh lý như biệt hóa, tăng sinh, apoptosis và phát triển. Tuy nhiên, nghiên cứu về sự điều hòa miRNA của ATG ở động vật giáp xác còn hạn chế.
Các nghiên cứu gần đây đã phát hiện ra rằng trong quá trình nhiễm virus hội chứng đốm trắng (WSSV) ở tôm đốm, miR-71 có thể ngăn chặn sự biểu hiện của gen peptide-1 (cap-1) liên quan đến vôi hóa của vật chủ, gây ra bệnh tự thực tế bào ở tôm và thúc đẩy sự nhân lên của virus trong cơ thể tôm. Hơn nữa, sự điều hòa miR-13b của knickkopf điều chỉnh chung cả tình trạng nhiễm WSSV ở tôm và mức độ tự thực bào trong tế bào vật chủ của tôm.
Tuy nhiên, với những mầm bệnh như Vibrio harveyi còn rất hạn chế. Do đó, gần đây, nghiên cứu của Chao Zhao và cs (2024) đã xác định các miRNA ứng cử viên đồng điều chỉnh PmATG5 và PmATG12, đồng thời giải mã vai trò điều chế qua trung gian miRNA của hệ thống liên hợp ATG5-ATG12 trong phản ứng tự thực đối với stress V. harveyi ở tôm sú. Bằng cách biểu hiện quá mức hoặc làm im lặng các miRNA này ở tôm sú, các tác giả đã phân tích các cơ chế mà hệ thống liên hợp ATG5-ATG12 điều phối phản ứng tự thực. Kết quả nghiên cứu này đã cung cấp những hiểu biết có giá trị về cơ chế phân tử phụ thuộc miRNA làm cơ sở cho phản ứng miễn dịch tự thực ở tôm và đưa ra các mục tiêu tiềm năng cho các liệu pháp phòng bệnh trong nuôi tôm.
Đặc tính phân tử và điều tra phát sinh gen của PmATG5 và PmATG12
Trình tự cDNA có độ dài đầy đủ của PmATG5 là 2281 bp, bao gồm 114 bp 5′UTR, 1357 bp 3′UTR và khung đọc mở (ORF) 810 bp mã hóa 269 gốc axit amin, tạo ra protein có cấu trúc lý thuyết trọng lượng phân tử là 31,125 kDa và điểm đẳng điện lý thuyết là 5,66 (Hình bổ sung S1a). Trình tự cDNA có độ dài đầy đủ của PmATG12 là 1101 bp, bao gồm 16 bp 5′UTR, 722 bp 3′UTR và ORF 363 bp mã hóa 120 gốc axit amin, tạo ra protein có trọng lượng phân tử theo lý thuyết là 13,59 kDa và điểm đẳng điện lý thuyết là 6.12.
Trình tự axit amin ATG5 của tám loài khác và trình tự axit amin ATG12 của sáu loài khác đã được sử dụng để xây dựng nhiều cách sắp xếp trình tự và cây tiến hóa Trong liên kết nhiều trình tự với các loài khác, PmATG5 có độ tương đồng cao nhất với cua xanh (CAJ31266.1), cao tới 87% và rất phù hợp với các loài khác. PmATG12 cũng cho thấy mức độ tương tự 60% và 71% với các loài khác. Những kết quả này cho thấy PmATG5 và PmATG12 được bảo tồn tiến hóa cao với các gen tương đồng khác. Cây phát sinh gen cho thấy PmATG5 và ghẹ xanh (C. sapidus) tập hợp thành một nhánh duy nhất, trong khi PmATG12 tập hợp thành một nhánh riêng biệt. Trong cây phát sinh gen ATG12, PmATG12 được nhóm với các loài côn trùng như Danaus plexippus, Bombyx mori và Biston betularia.
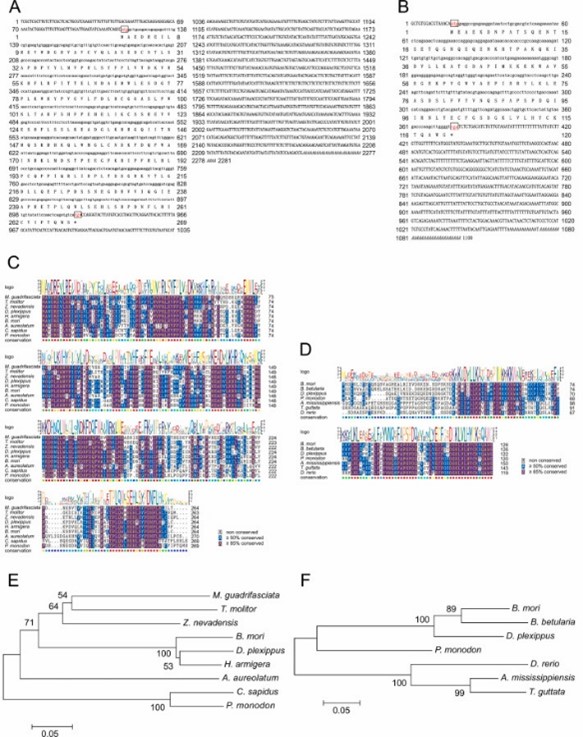
Phân bố mô của biểu hiện PmATG5 và PmATG12
Để nghiên cứu sự phân bố đặc hiệu mô và các kiểu biểu hiện mRNA của PmATG5 và PmATG12 ở tôm, các mô khác nhau, bao gồm gan tụy, cơ, ruột, dạ dày, cơ quan bạch huyết, mang, não và tim đã được kiểm tra. Cấu hình biểu hiện của PmATG5 (Hình 1a) và PmATG12 (Hình 1b) cho thấy cả hai đều biểu hiện mức độ biểu hiện cao nhất trong mô cơ, tiếp theo là gan tụy, ruột, mang và các mô khác. Đáng chú ý, biểu hiện PmATG5 là thấp nhất trong máu, trong khi PmATG12 biểu hiện tối thiểu ở tim.

Hình 1. Cấu hình biểu hiện đặc hiệu mô của các gen liên quan đến quá trình tự thực (ATG) P. monodon ATG5 (PmATG5) và ATG12 (PmATG12)
Phân tích tin sinh học của miRNA nhắm mục tiêu PmATG5 và PmATG12
Để khám phá tác dụng đồng điều hòa của miRNA đối với PmATG5 và PmATG12, việc sàng lọc ban đầu đối với miRNA có thể cùng điều chỉnh cả hai gen đã được tiến hành bằng phần mềm tin sinh học. Phân tích tin sinh học cho thấy một loạt miRNA có khả năng liên quan đến việc điều chỉnh ATG ở tôm sú. Cụ thể, 22 miRNA được xác định là bộ điều chỉnh tiềm năng của PmATG5 và 24 miRNA có khả năng điều chỉnh PmATG12. Đáng chú ý, ba miRNA—miR-7562, miR-8485 và miR-278—nổi lên như những ứng cử viên đồng điều chỉnh PMmATG5 và PMATG12 (Bảng 1). Hình 2 hiển thị các vị trí liên kết cụ thể nơi ba miRNA này có thể tương tác với 3′UTR của PmATG5 và PmATG12.
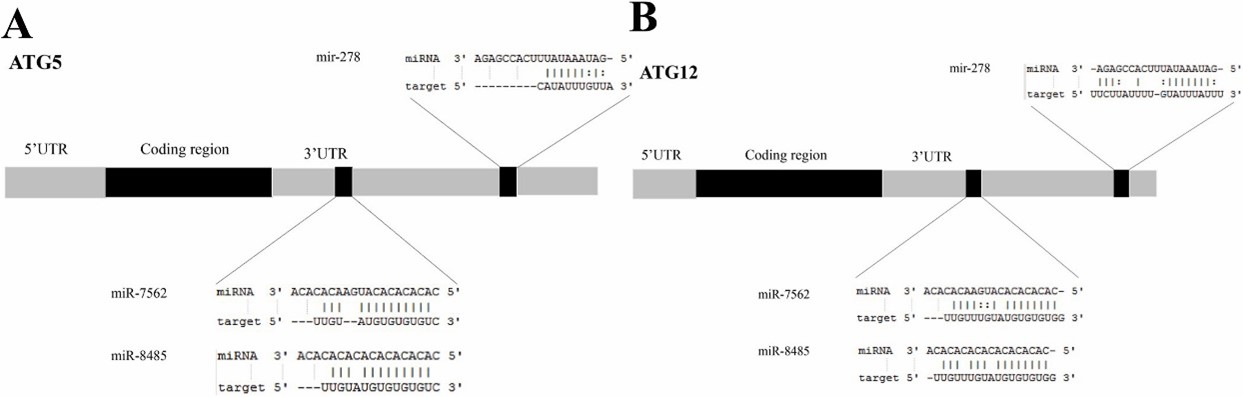
Hình 2. Sơ đồ biểu diễn các vị trí mục tiêu của miRNA trong 3’ vùng chưa được dịch mã (UTR) của các gen liên quan đến quá trình tự thực của tôm ATG5 (PmATG5) và ATG12 (PmATG12)
Sàng lọc các miRNA tác dụng chính và phân tích biểu hiện PmATG5 và PmATG12 sau khi nhiễm khuẩn
Để hiểu tác động điều chỉnh của ba miRNA đối với sự biểu hiện của PmATG5 và PmATG12, qRT-PCR đã được sử dụng để đánh giá mức độ biểu hiện gen và miRNA sau khi nhiễm V. harveyi. Các xét nghiệm qRT-PCR cho thấy các mẫu biểu hiện đặc biệt sau nhiễm trùng (Hình 3). Hình 3a cho thấy biểu hiện miR-278 đã được điều chỉnh giảm đáng kể ở cả 6 và 24 giờ sau khi bị nhiễm bệnh. Một xu hướng tương tự đã được quan sát đối với miR-7562, cho thấy sự biểu hiện giảm đáng chú ý tại cùng thời điểm (Hình 3b).
Ngược lại, mức độ miR-8485 không thay đổi sau khi bị lây nhiễm (Hình 3c). Đối với các gen mục tiêu quan tâm, dữ liệu qRT-PCR cho thấy sự gia tăng tạm thời trong biểu hiện của PMATG5, với mức mRNA tăng khoảng 1,8 lần sau 48 giờ và 3,8 lần sau 72 giờ sau nhiễm trùng, so với các đối chứng không bị nhiễm bệnh ( Hình 3d). Tương tự, biểu hiện PmATG12 tăng đáng kể sau 6 giờ sau khi nhiễm bệnh, đạt cực đại ở 72 giờ, với mức biểu hiện cao hơn khoảng 1,6 lần so với mức đối chứng (Hình 3e).

Hình 3. Sự biểu hiện khác biệt của microRNA (miRNA) liên quan đến các gen liên quan đến quá trình tự thực của tôm sú (ATG) ATG5 (PmATG5) và ATG12 (PmATG12)
Phân tích trình báo cáo Dual-luciferase để xác nhận các bộ điều chỉnh miRNA của PmATG5 và PmATG12
Để xác thực tác dụng điều hòa của miR-278 và miR-7562 đối với 3′UTR của gen mục tiêu ATG5 và ATG12, các tác giả đã sử dụng xét nghiệm báo cáo luciferase kép. Kết quả trong Hình 4a chứng minh rằng miR-278 có thể làm giảm đáng kể biểu hiện luciferase của ATG5 3′UTR (p < 0,0001); tuy nhiên, sau khi thay đổi vị trí gắn kết, mối quan hệ điều hòa vẫn không thay đổi (p = 0,5071). Ngược lại, như trong Hình 4b, miR-7562 có thể điều chỉnh biểu thức luciferase liên quan đến ATG5 3′UTR (p < 0,0001);
Sau đột biến vị trí liên kết miRNA trên ATG5 3′UTR, tác dụng điều hòa này bị suy giảm 9,3 % (p = 0,0022), tuy nhiên miR-7562 vẫn duy trì ảnh hưởng điều hòa lên sự biểu hiện luciferase ATG5 3′UTR của ATG5 3 bị đột biến `UTR (p = 0,0022). Các kết quả được mô tả trong hình 4c chỉ ra rằng miR-278 có thể điều chỉnh biểu hiện luciferase của ATG12 3′UTR (p < 0,0001). Sau khi đột biến vị trí liên kết miR-278 trong ATG12 3′UTR, tương tác điều hòa này đã giảm đáng kể 25,4% (p < 0,0001), mặc dù miR-278 tiếp tục điều chỉnh biểu hiện luciferase của ATG12 3′UTR bị đột biến (p < 0,0001).
Ngoài ra, Hình 4d cho thấy biểu hiện luciferase được điều chỉnh bởi miR-7562 được liên kết với ATG12 3′UTR (p < 0,0001). Sau khi làm thay đổi vị trí liên kết miR-7562 trong ATG12 3′UTR, mối quan hệ điều hòa này đã bị suy yếu đáng kể 46,2%, tuy nhiên miR-7562 vẫn tiếp tục thể hiện tác dụng điều chỉnh đối với biểu hiện luciferase của ATG12 3′UTR bị đột biến (p = 0,0246).
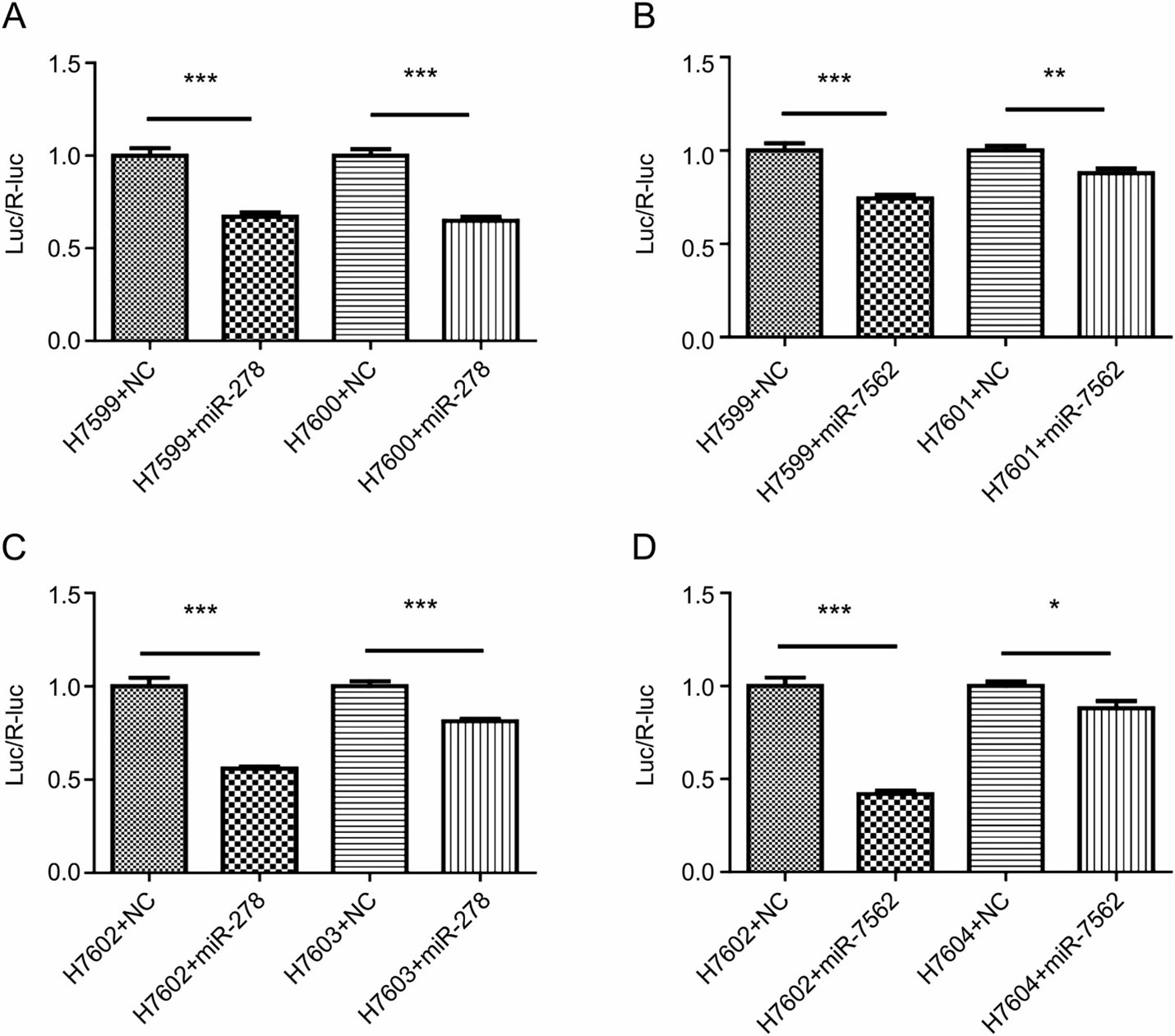
Hình 4. Phân tích sự tương tác giữa các microRNA khác nhau (miRNA) và 3’ vùng chưa được dịch mã (UTR) trong các gen liên quan đến quá trình tự thực (ATG) P. monodon ATG5 (PmATG5) và ATG12 (PmATG12) bằng cách sử dụng trình báo cáo luciferase kép xét nghiệm.
a ) Phân tích liên kết của miR-278 với PMATG5 3′UTR. (b) Phân tích liên kết của miR-7562 với PmATG5 3′UTR. (c) Phân tích liên kết của miR-278 với PMATG12 3′UTR. (d) Phân tích liên kết của miR-7562 với PmATG12 3′UTR. những thay đổi trong tín hiệu luciferase là dấu hiệu cho thấy khả năng nhắm mục tiêu của miRNA đối với 3′UTR của PmATG5 và PmATG12.
Điều chế biểu thức PmATG5 và PmATG12 và khả năng tự thực của miR-7562
Để nghiên cứu khả năng điều tiết của miR-7562 trên PmATG5 và PmATG12, ảnh hưởng của miR-7562 đến mức độ phiên mã của các gen này được đánh giá bằng cách làm im lặng và điều chỉnh tăng biểu hiện miR-7562. Phân tích qRT-PCR cho thấy sự biểu hiện quá mức của miR-7562 đã ức chế đáng kể mức độ phiên mã PmATG5 xuống lần lượt là 0,67 và 0,81 lần so với đối chứng ở thời điểm 24 và 48 giờ sau điều trị (Hình 5a).
Ngược lại, việc giảm miR-7562 dẫn đến sự gia tăng đáng kể PmATG5 mRNA, đạt gấp 2,2 lần so với đường cơ sở sau 24 giờ. Tương tự, sự biểu hiện quá mức của miR-7562 dẫn đến giảm rõ rệt PmATG12 xuống 0,62 lần so với nhóm kiểm soát sau 24 giờ, trong khi miR-7562 làm im lặng biểu hiện PmATG12 tăng lên 1,43 lần so với mức kiểm soát (Hình 5b). Việc điều chế quá trình tự thực bằng miR-7562 đã được nghiên cứu sâu hơn bằng cách phân tích mức protein bằng phân tích Western blot, sử dụng tỷ lệ LC3I/LC3II làm thước đo hoạt động tự thực. Sự biểu hiện quá mức của miR-7562 khi sử dụng cấu trúc miR-7562-agomir đã làm tăng đáng kể tỷ lệ LC3I/LC3II sau 48 giờ sau can thiệp so với nhóm đối chứng (Hình 5c). Ngược lại, sự ức chế miR-7562 bằng cấu trúc antagomir làm giảm đáng kể tỷ lệ LC3I/LC3II.
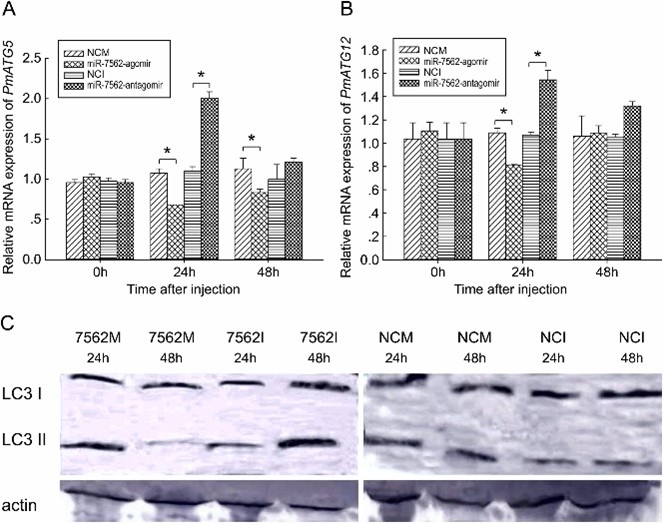
Hình 5. Tác dụng của miR-7562 đối với sự biểu hiện gen liên quan đến quá trình tự thực và hoạt động tự thực
(b) Biểu thức thay đổi của PMATG12 sau khi biểu hiện quá mức hoặc im lặng có mục tiêu của miR-7562. (c) Ảnh hưởng của miR-7562 đến tỷ lệ tế bào chất (LC3-I) của protein liên kết với vi ống 1A / 1B-chuỗi ánh sáng 3 (LC3-I) đến bị mất hiệu lực (LC3-II) khi miR-7562 bị biểu hiện quá mức hoặc im lặng. Mức độ biểu hiện được định lượng bằng phương pháp 2 −ΔΔCt.
Tóm lại, đây nghiên cứu đầu tiên nhân bản và mô tả các ATG chính, PmATG5 và PmATG12, ở tôm sú và miR-7562 được xác định là miRNA tiềm năng điều chỉnh cả hai gen này. Các thí nghiệm in vivo đã chứng minh rằng miR-7562 điều chỉnh quá trình tự thực bằng cách điều chỉnh tiêu cực hệ thống liên hợp ATG5-ATG12 trong quá trình nhiễm V. harveyi. Những phát hiện này lần đầu tiên tiết lộ mối liên hệ điều tiết giữa miR-7562 và bộ máy tự thực ở tôm sú, nêu bật tiềm năng của nó như một bộ điều biến chính của phản ứng miễn dịch của vật chủ.










_1769663223.jpg)

_1769399561.jpg)
_1762138517.jpg)







